
撰文:高子阳、Verdi
突破技术
这是人体中各种细胞类型的完全目录。
重要意义
超精确的人类生理学模型将加速新药的研发与试验。
技术成熟期
5年
主要研究者
-布罗德研究所(Broad Institute)
-桑格研究所(Sanger Institute)
-陈-扎克伯格的Biohub(Chan Zuckerberg Biohub)
当你凝视脚下的方寸土地,是否曾经好奇过生命的起源?鹰击长空,鱼翔浅底,万类霜天竞自由,这一切都源于35亿年前混沌海洋中的生命微光。当时的地球表面遍布着频繁活动的火山,向外不断地喷吐出火山灰和岩浆;空气中的一氧化碳和氢气在电离子风暴的作用下形成了简单的有机物,这些物质在原始海洋中不断地堆积,在洪荒之初发生了复杂的化学反应,构成了生物大分子,逐渐演变成最初的生命。
昔日原始海洋中的沧海一粟,是如何变成今日的勃勃生机的呢?通过化石记录的证据,我们可以确定生命起源于35亿年前的原核细胞生物。在生命出现后的最初的15亿年间,原核细胞是唯一的生命形式,它们个体渺小但数量巨大,有些通过光合作用产生氧气,彻头彻尾地改变了地球的模样。原核细胞并没有细胞核,构成其核物质的是直接与细胞质接触的单个染色体。我们熟悉的蓝藻、各类细菌都是原核生物,构成这种古老生命形式的原核细胞,依旧渗透在我们生活的每一个部分。
大约在21亿年前,原始海洋中的有机物逐渐被消耗殆尽,大气中积攒了部分原核生物通过光合作用产生的氧气,原始海洋中逐渐演化出真核细胞。与原核细胞不同的是,真核细胞的内部具有核膜包被的细胞核。最初的真核细胞是单细胞的真核生物,又叫原生动物,仅一个小小的细胞就具有完整的生命代谢体系。后来,细胞之间产生了分工与合作,不同的细胞各司其职,进化出了多细胞生物。植物、动物、真菌等均属于多细胞的真核生物。
与细胞的漫长进化史相比,人类在地球上生活的时间可谓流光瞬息。尽管我们使用高级的工具改变了地球的环境,建立了发达的文明,但人类的本质是一种灵长目人科人属的直立行走物种。人类是多细胞生物,人体内的细胞之间相互协作,通过发生惊人的化学反应,使我们的生命得以延续。
而我们在很长的一段时间内,对此一无所知。
人类对细胞的认识,起源于400年前。
人们假想的含有“小矮人”的精子(Nicholas
Hartsoeker,1695年)

Robert Hooke通过显微镜看到并画下了软木栓细胞(1665年)
1665年,罗伯特·胡克使用自制的光学显微镜观察软木薄片时,观察到了像修道院的房子一般的一个个小隔间。于是他使用表达单人间意思的cell一词将植物细胞命名为cellular。尽管胡克观察到的细胞早已死亡,其结构实为死亡后的植物细胞壁,但胡克仍然被认为是第一个描述细胞结构的科学家[1]。在罗伯特·胡克观察到死细胞后不久,1674年,荷兰科学家列文虎克利用手工自制的显微镜,首次观察到了雨水中活动的单细胞生物,这是人类历史上第一次成功地观察并描述了活细胞[2]。
19世纪初期,随着植物解剖学的发展,法国科学家Charles Milbel认识到植物的每一个部分都有细胞存在。随着种种观察的累积,1838年法国科学家施莱登宣布细胞是一切植物的基本活体单位,是一切植物发展的根本实体。1839年,施旺将此学说扩大到动物界,如此便构成了人们所熟知的细胞学说:细胞是动物和植物生命活动的基本单元。恩格斯将细胞学说誉为19世纪的三大发现之一,是整个现代生物学的理论基础[3]。
随后的200年中,科学家对细胞学说进行了不断的改进和纠正,逐渐发展为现代细胞学说。现代细胞学说主要分为三点:1.细胞是一个有机体,一切动植物都由细胞发育而来,并由细胞和细胞产物所构成;2.细胞是一个相对独立的单位,既有自己的生命活动,又对其他细胞和其他细胞共同组成的生命整体起作用;3.新的细胞是从以前存活的细胞中产生的。
1873年,意大利组织解剖学家高尔基发明了“高尔基银染法”,使得人们可以看到完整的神经细胞。1887年,西班牙画家拉蒙·卡哈尔利用此方法对小脑和视网膜进行观察,并通过其出色的绘画才能,精确地呈现出他的研究结果。卡哈尔观察到小脑中的不同的神经细胞通过突起的方式相互勾连,但并不直接连接[4]。1889年,卡哈尔发表了自己的研究成果,他提出大脑是由相互独立的神经细胞所构成,它们之间并不构成直接相连的网状系统。这便是神经元学说的基本内容。
植物细胞(J.M.Schleiden,1838年)
此外,卡哈尔发现神经细胞并不是一模一样的,于是他通过其画作展示了人脑中不同的神经细胞类型,如锥体细胞、浦肯野神经元等。在卡哈尔笔下的视网膜结构图中,他将视网膜分为10层,每层由不同种类的细胞组成。卡哈尔认为B、C、D层的感光细胞将光线转换成电信号,之后传导至其他细胞层,最终传导至大脑。1906年,瑞典卡罗琳斯卡医学院将诺贝尔生理学或医学奖授予在神经组织学领域做出重要贡献的高尔基和卡哈尔。
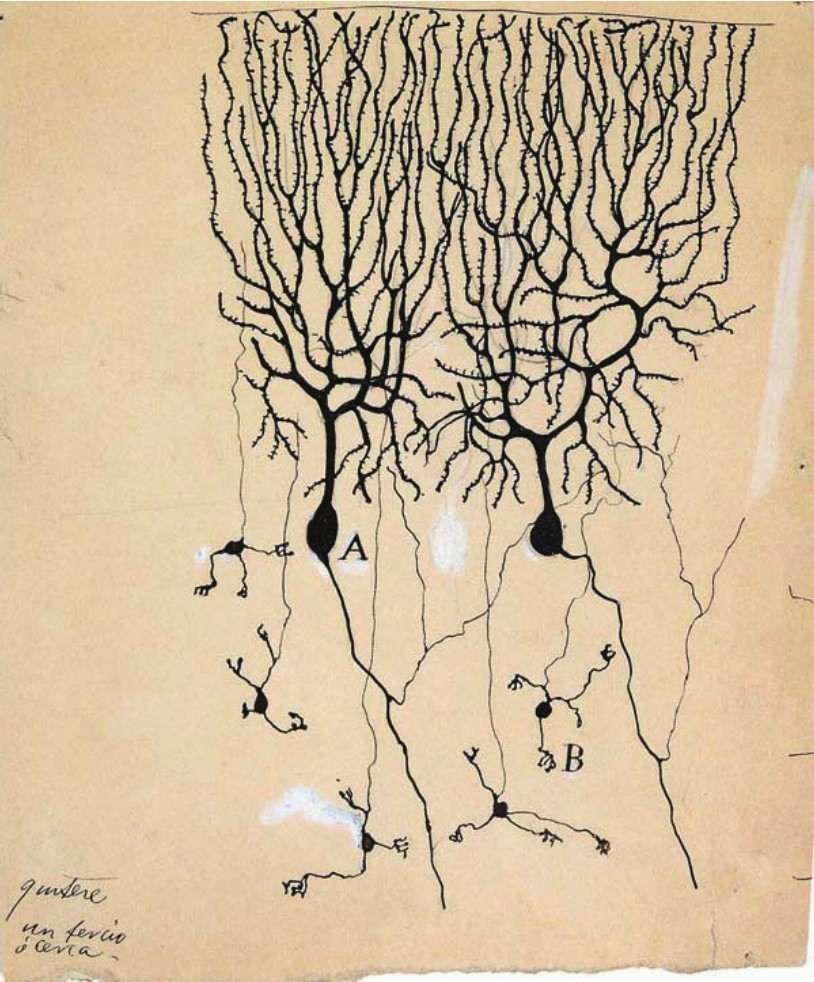
神经细胞(Roman y Cajal)
今天,在卡哈尔绘制精细的细胞图谱100年后,我们已经知道,除红细胞外,人体内拥有总数超过1万亿的细胞。它们分布于人体的每一个部分,行使各自不同的功能,构成了精妙绝伦的人体结构,每时每刻满足着新陈代谢的需要。目前人们对细胞分类的标准主要是基于细胞的形态、基因表达和功能的差异,细胞的空间分布、分化状态和谱系追踪对于细胞分类起着辅助的作用。近年来,随着表观遗传学研究的深入,表观遗传谱也成为细胞分类学关注的重点。如果只进行粗略的分类,人体内大概有300种细胞类型。但是如果进行精细的划分,光是视网膜神经元就有100多种细胞类型,由此可见人体内的细胞构成极其复杂。
神经细胞(A.von Kolliker,1852年)
拿人体内最大的器官——皮肤为例。看似简单的皮肤组织,实际上是由许多种细胞组成的。人类的皮肤分为表皮和真皮两层,被表皮基底层分隔开。表皮是皮肤的最外层,是人身体的保护层,具有维持水分、避免病原菌进入体内的功能;皮肤表皮中具有柱状上皮细胞、黑色素细胞、角质细胞、扁平细胞等细胞[5]。真皮是位于表皮以下的组织,由致密的结缔组织组成,其中含有大量的成纤维细胞、肥大细胞、组织细胞、淋巴细胞、噬黑色素细胞、朗格汉斯细胞及少量的真皮树突状细胞[6]。
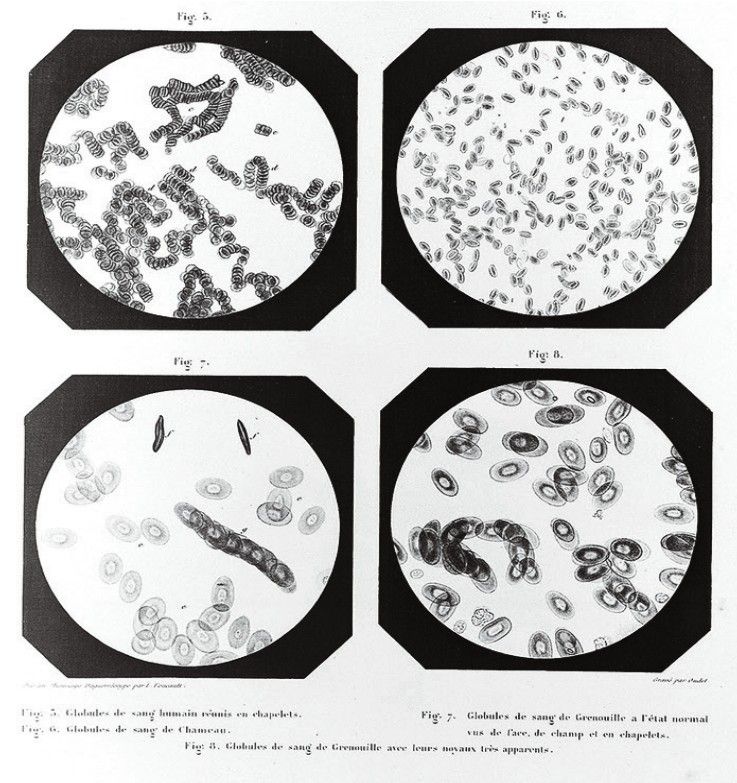
人类、骆驼和蟾蜍的血液细胞Daguerreotypes(A.Donné,1845年)
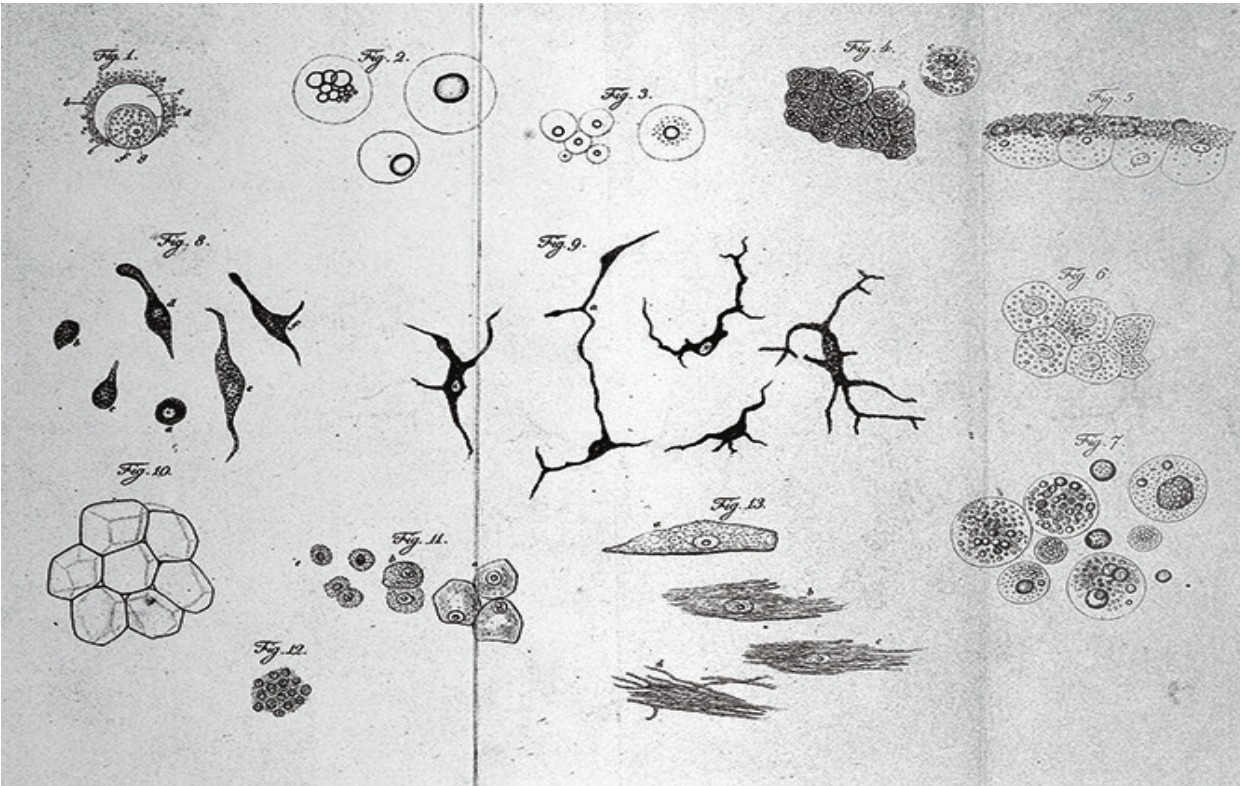
动物细胞草图(Theodor Schwann,1839年)
皮肤拥有很强的修复和再生能力,这是由于皮肤中的皮肤干细胞在起作用。目前研究比较多的是表皮干细胞和毛囊干细胞。表皮干细胞是各类表皮细胞的祖细胞,可以向下迁移分化成表皮基底层,进而发育为毛囊,也可以向上迁移,最终分化为各类表皮细胞[7]。仅仅皮肤干细胞的类型就有很多。在胚胎时期,皮肤是由在毛囊形成过程中不同的细胞类型所构成的。科学家们通过对胚胎皮肤毛囊祖细胞进行转录组图谱的分析,鉴定出简单的毛囊祖细胞可以被分为6种类型,它们在细胞黏附、细胞迁移、上皮发育等过程中都扮演着各自不同的角色[11]。表皮干细胞可以被不同的生物标记物区分为6种类型,它们呈片状分布在表皮基底层[8]。
成纤维细胞是一种合成细胞外基质和胶原蛋白的细胞,是生物结缔组织的基本构造,为结缔组织提供框架结构,对皮肤的伤口愈合有着重要作用[9]。看似简单的成纤维细胞也各自不同,可以根据其发育的不同阶段分为7种类型,每种成纤维细胞的形态各异,其所处位置和活动性决定了细胞的形态[10]。
另外,人类皮肤中还包含广泛的免疫细胞网络,是皮肤免疫屏障的关键。人们利用皮肤免疫细胞的细胞图谱分析,发现了皮肤免疫细胞的功能、位置的异质性。一些特殊类型的T细胞仅分布于耳周皮肤,具有不同免疫应答功能的肥大细胞也具有不同的数量[12]。
再拿卡哈尔研究过的视网膜举个例子。视网膜是脊椎动物和一些头足纲动物眼球后部的一层非常薄的细胞,行使将光信号转化为神经信号的功能。它的结构清晰明了,是很多科学家喜爱的研究对象。人类的视网膜由外到内分为10层,每层都由特异的细胞构成。以往估计人体内总共有300多种细胞,而现在人们发现单单一层薄薄的视网膜就可能含有100多种神经细胞。通过对转录组的分析,研究者发现了几十年的眼部研究都没能发现的两种新类型的视网膜细胞。
简单的视网膜细胞种类就已经大大超出了人们的想象,更不用说复杂的人脑了。人脑中同样具有高度异质的细胞类型,主要包括神经元和神经胶质细胞。神经胶质细胞中的寡突胶质细胞从转录组就能分出多于12种的类型。神经元细胞在转录组层面具有高度异质性,即使是形态无差异的神经元,其中的蛋白表达也存在着很大的变化。
这些例子告诉我们,传统的基于细胞形态的分类学对于细胞类别的鉴定仅停留在表面阶段,人类对构成自己身体的细胞的种类了解甚少。作为第一个描述细胞的科学家,胡克将会被生物学的下一个大型项目震惊到:这是一个使用现代基因组学和细胞生物学中最强大的工具来单独捕获和端详数百万个细胞的计划。
这个项目的目标是构建第一个全面的细胞分类系统——“细胞图谱”或者“人类细胞地图”。这个项目的实现将成为一个技术奇迹,因为它将首次全面揭示人体是由什么组成的,并为科学家们提供一个新的复杂的生物学模型,以提升药物研发的速度。
这项研究主要面临着两个挑战。一是细胞分类的准确性。细胞分类系统需要能够定义绝对单一的细胞类型,而不是定义一个具有多种细胞类型的亚群。二是细胞分类的全面性。我们需要的是一个能够保证最终鉴定出所有细胞类型的分类系统,这同样要求我们能够找到所有的细胞类型[13]。
为了实行这个解码人体37.2万亿个细胞的任务,由来自美国、英国、瑞典、以色列、荷兰和日本的国际科学家组成的联合会正在分配任务,包括检测每种细胞的分子特征,并给每种细胞一个在人体空间中特定的“邮政编码”。“我们将会看到我们所期望的东西、我们已知存在的东西,但我确信除此之外我们还会发现全新的事物。”英国桑格研究所的细胞图谱团队负责人Mike Stubbington说,“我认为会有惊喜出现。”
这个新项目的研究主要运用了3种技术:单细胞捕获技术、测序技术和多种全新的标记和染色技术。前两项技术通常一起讨论,称为“单细胞测序技术”。
单细胞测序技术
单细胞RNA(核糖核酸)测序是细胞图谱计划的核心技术,能够提供单个细胞的基因组图谱。而且目前大量的与之相配的技术,如微流控等使单细胞测序成为一种低成本、高通量的测序方式[14,15]。
在单细胞RNA测序技术出现之前,人们普遍利用分子探针型技术对单细胞进行m RNA(信使核糖核酸)图谱的分析。常见技术主要有荧光融合报告蛋白、定量实时聚合酶链式反应(q RT-PCR)、原位荧光杂交 (FISH)和基因芯片技术[19]。这些技术在一定层面上可以反映细胞中的一些基因的表达变化,但由于它们都是依赖于分子探针的技术,并不能够反映出全基因组的转录组水平,空间分辨率较低,具有一定的局限性。
单细胞RNA测序是一种不需依赖分子探针的技术,它将细胞内的RNA分子反转录成c DNA (互补脱氧核糖核酸),并随之进行二代测序,能够反映出全基因组层面的编码和非编码RNA转录水平,是反映特定环境中细胞状态的绝佳方法。
单细胞RNA测序最早是由大规模(bulk) RNA测序演变而来的。对于大规模RNA测序而言,提取RNA时大概需要100万个细胞。
而单细胞测序需要使用单细胞捕获技术,对单个细胞进行裂解,然后测序[20]。2009年,M.Surani等人在《自然·方法》杂志上发表了多聚A尾法的单细胞RNA测序方法,他们利用带多聚T尾的引物对模板进行两次扩增后得到c DNA,这项方法在后来的单细胞RNA测序研究中得到了非常广泛的应用[21]。
近年来,单细胞RNA测序更是飞速发展:2012年,Gary Schroth和Richard Sandberg等人在《自然·生物》技术杂志上发表了名为Smart-seq的单细胞测序方法,这项方法利用了模板转移的技术,提高了转录过程的覆盖度[22];2011年,James Eberwine等人将体外转录技术(IVT)用于单细胞RNA测序,实现了c DNA的线形扩增[23];同样在2011年,Tung T.Hoang等人发表了滚动循环周期的单细胞RNA测序技术,这使得对原核生物(细菌、放线菌等)进行单细胞RNA测序成为可能[24];2012年,Peter Lonnerberg和Sten Linnarsson等人发明了一种对单细胞进行RNA 5’端测序的方法,这种方法满足了对特定核酸链进行测序的需求[25];2012年,Itai Yanai等人发明了CEL-Seq的单细胞测序方法,通过汇聚标记过后的样本,满足了单细胞RNA的快速线形扩增[26];2013年,Gosta Winberg和Rickard Sandberg在Smart-Seq的基础上进行了改进,推出了Smart-Seq2技术,在单细胞的反转录、模板转移、预扩增方面都有了显著的提高。与Smart-Seq相比,Smart-Seq2有着更精确的检测灵敏度、更高的测序覆盖率[34];2014年,来自Weizmann研究所的Ido Amit和Amos Tanay发明了一种自动化的高通量单细胞RNA测序的并行技术,能够在数以千计的单细胞中分析转录组状态。利用这项技术,人们可以从复杂的组织中测得广泛的细胞组成类型[16]。
单细胞测序的流程主要分为单细胞捕获、单细胞裂解、反转录、核酸扩增、建库测序这5个步骤。单细胞捕获是单细胞测序的第一步,这看似简单的步骤实际上是整个测序过程中最具有挑战性的一步。
目前,最常用的单细胞捕捉技术有微管吸吮技术、激光显微切割、荧光活化细胞分选、微滴技术、微流体技术。微管吸吮技术采用开口极细的微型玻璃管,利用压力将单个细胞从组织中捕获,这是一种十分耗时的方法,被用于早期胚胎的单细胞捕获。激光显微切割是利用激光将组织薄片上的细胞进行分离的技术,同样非常耗时,被用于基于细胞形态和细胞荧光标记的细胞分选。
为了提高细胞分选的效率,人们开始尝试用高通量的方式。荧光活化细胞分选(FACS)是一种利用细胞表面的荧光标记对液滴中的单细胞进行分离的技术,其优点是高通量,且能够利用特定免疫标记的细胞表面分子提高分选的准确性。但其缺点也很明显,一是需要特定的抗体和分子标记,二是仪器的价格昂贵[29]。
微流体技术是利用微流体芯片在极窄的流体通道中将细胞进行分离的方法,它能够分离总体积量很小的细胞,同样是一种高通量的方法。Fludigm C1的微流体平台能够利用流体回路对细胞进行捕捉,同时能用显微镜对这些细胞进行观察,反转录和核酸扩增都自动紧随细胞捕捉后进行。但是微流体技术要求细胞的大小基本一致,在处理黏性较高的非球体细胞时,效率也会降低。它同样也是一种高成本的方法[30]。
激光显微切割是一种在显微镜下利用激光将单个细胞从组织薄片中切除的技术,可以看作是一种物理分选,它能够保留细胞的周围信息。但是如此小范围的精细切仍然是一个技术上的挑战,而且激光切割过程中的紫外线可能会对细胞具有潜在的伤害[31]。目前并未有一种能适用于所有研究的单细胞分离方法,研究者需要根据其研究目的和样本的种类选择最适合实验的方法。
2015年,一种叫作“细胞微流体”(Drop-Seq)的技术进入了人们的视线。Drop-Seq是一种从液滴中提取单个细胞进行转录组分析的方法[27]。它利用微流体装置来区分转录后的单个细胞、裂解液和覆盖着引物的微珠。这项技术分离单独的细胞并用微珠标记,使其被油滴包裹后再进行研究和分析。选择油滴的原因是油滴可以如同汽车一样载着细胞,沿着被蚀刻在微小芯片上的狭窄的毛细管单向“街道”分流,使得细胞被聚集在特定的地方,裂解并逐一研究。细胞微流体是一种低成本高通量的测序方法,其成本大约为每个细胞7美分,是很多研究者的首选。但是它对于微流体设备的要求较高,对于不同的细胞需要特定种类的微流体设备来进行液滴的分离。另外Drop-Seq对于单个细胞的基因敏感度较低,且只能用于m RNA的反转录[28]。
单细胞分选的下一步是裂解细胞,然后对细胞中的多聚A尾RNA进行反转录处理。反转录时,我们以RNA为模板,利用多聚T的引物进行首链的合成,并且在合成的序列后添加上基因条形码,以便后续的分析识别。次链的合成有c DNA合成和模板转移扩增两种方式,最终都可以得到双链的DNA。

细胞微流体技术使用的微流体设备
再下一步是对反转录后的DNA进行核酸扩增。一种是利用聚合酶链式反应(PCR)的扩增方法,这是指数型的扩增;另一种是体外转录法,这种方法是线性的扩增,但需要对RNA进行多一轮的反转录。完成反转录后便可以对扩增后的DNA文库进行测序。
单细胞RNA测序目前已被广泛应用于检测组织中的复杂的细胞种类、追踪细胞谱系的来源、检测细胞生理状态等方面,大大助力于各项研究:2010年,来自剑桥大学的M.Surani对囊胚期中不同发育阶段的细胞进行了单细胞RNA测序,发现了在这两种状态下细胞的转录水平存在巨大的差异,大多与对总体代谢有重大影响的分子有关[18];2016年,来自斯坦福大学的Marius Wernig和Stephen Quake利用单细胞RNA测序技术在不同的时间点对小鼠胚胎成纤维细胞到诱导神经细胞的重编码过程进行了分析,解释了细胞重编码过程中分子的连续性。这项研究对于理解在分化过程中的细胞转录组状态具有重要的意义[17]。
目前而言,单细胞RNA测序的技术已经被广泛运用于真核生物的多聚A尾m RNA的转录组研究中,但是仍然有很多问题需要解决。比如在进行短序列测序时,很难同时做到维持核酸链的特异性和检测各类亚型之间的差异。在测序过程中,RNA的丢失(50%~60%)会在很大程度上降低转录组分析的全面性[32]。单细胞RNA测序的敏感度同样也是目前单细胞测序的不足之处,目前在低量的转录组中,很难辨别出技术噪声和生物差异性之间的区别,这使得人们在研究整体的转录水平时会损失很大一部分信息[33]。此外,对于非真核细胞的单细胞测序研究,比如研究某些具有感染性的病原体,也需要人们在现有的单细胞测序技术上进行改进。
近些年关于单细胞RNA测序的研究使我们了解到,很多细胞层面的未解之谜只能通过单个细胞的研究来回答。我们可以想象,在不久的将来,当人们改进了这些技术性问题后,单细胞RNA测序的技术可以被用于所有类型的细胞转录组研究,人们也将揭示更多的单细胞层面上的未知问题。
单细胞RNA测序的另一个核心技术就是测序技术。测序是指通过物理或化学的方法确定线状生物大分子初级结构的过程。DNA测序指分析特定DNA片段的碱基序列,即腺嘌呤(A)、胸腺嘧啶(T)、胞嘧啶(C)和尿嘧啶(G)的排列方式。它是现代测序技术的核心,也是破解各种生物奥秘的关键。
20世纪中期,DNA测序技术刚刚起步,当时所流行的化学降解法、双脱氧链终止法、荧光自动测序、杂交测序等被统称为第一代测序。其中由Fred Sanger及其同事发明的双脱氧终止法(又称Sanger测序法)是第一代测序中最常被使用的技术。
双脱氧终止法(Sanger测序法)的原理是DNA复制,起反应体系中包括目标DNA片段、脱氧三磷酸核苷酸(d NTP)、双脱氧三磷酸核苷酸(dd NTP)、测序引物及DNA聚合酶等。由于dd NTP缺少3’-OH基团,不具有和另一个d NTP连接形成磷酸二脂键的能力,可以终止DNA链的延伸。通过在4个平行的测序反应中分别加入不同的dd NTP,DNA链会分别在A、G、C、T位终止,于是会形成不同长度的DNA片段。随后通过聚丙烯酰胺凝胶电泳区分开长度相差为一个核苷酸的DNA分子,于是便可以读出DNA序列[35]。
2000年,人类基因组计划的草图完成了。传统的第一代测序已经不能满足对大规模基因组进行测序的需求,此时新一代测序的技术在传统科学和商业界的推动下应运而生。
第二代测序的核心技术是边合成边测序,即通过捕捉新合成的核酸末端的标记来确定DNA的序列,与Sanger测序法相比具有更快的测序速度。第二代测序技术最显著的特征是高通量,能够一次性对上百万条DNA进行测序,使DNA测序的成本降低到了以前的千分之一。采用大规模平行测序平台的第二代测序技术,打破了以往大型测序中心对测序产业的垄断,使DNA测序费用降到了以往的百分之一。第二代测序技术的发展使人们能用低廉的价格更加全面地研究基因组、转录组、表观遗传等组学之间的关系。目前市面上主要的第二代测序平台有罗氏公司的454焦磷酸测序、Illumina公司的Hi Seq和Life Technologies公司的SOLi D。Illumina是单细胞RNA测序最常使用的平台,整个测序分为4个步骤:文库制备、核酸簇生成、DNA片段测序、数据分析[36]。
近几年来,第三代测序技术的发展势头十分猛烈,与第二代测序技术不同的是,第三代测序技术不需要进行PCR扩增。目前盛行的第三代测序技术有Helico Bio Science的单分子测序技术、Pacifc Bioscience的SMRT技术和Oxford的Nanopore(纳米孔单分子测序)技术。Nanopore作为最常用的第三代测序技术,与其他利用“边合成边测序”原理的技术都不同。该公司利用一种经过特殊设计的纳米孔,将核酸外切酶依附在孔的外表面,将一种合成的环糊精通过共价键安装在孔的内表面,充当传感器的角色。当DNA分子从孔中经过时,会使流经纳米孔的电流强度发生变化,再利用灵敏的电子设备检测到这些变化,从而鉴定出这些碱基[37]。
对于测序而言,测序技术的应用只是其得到序列数据的一种方式,在得到数据后更重要的步骤是对这些数据进行分析且得出对实验有用的结论。无论是大规模细胞还是单细胞的转录组测序,目前其分析的流程都大同小异。
第一步都是序列比对和了解片段的测序深度。所谓序列比对就是将测序后的结果与已有数据库中的模板序列进行比对,再将与之匹配的模板序列的模板信息安置到测序后的序列上。第二步是要对比对后的序列进行质量控制,主要关注基因文库的质量是否能够满足后续分析的需要;在单细胞RNA测序中,还需要关注单个细胞的RNA是否被降解。第三步是在确定得到质量过关的测序数据后,对测序的深度进行标准化处理,以确保在分析时不同批次的数据具有相近的测序深度。以上步骤我们称之为测序数据的上游分析。
下游的分析在大规模细胞测序和单细胞测序中并无差异。首先我们需要对令人疑惑的因素进行分析,利用回归分析的手段找出细胞或样本之间的潜在差异。其次便可以用聚类分析的手段对细胞类型进行鉴定。最后利用差异表达分析工具分析不同细胞类别的特征。得到了以上步骤的结果后,我们便可以进行更加复杂的分析和模型构建,如基因调控网络分析、单细胞的转录动力学分析等[38]。
细胞图谱的绘制不仅需要强大的单细胞测序技术,还需要依靠多种全新的标记和染色技术来明确特定细胞的空间坐标。近年来,激光显微切割和原位荧光杂交技术也在趋于单细胞化,大大提高了空间分辨率。而组织透明化技术为在组织中精确定位细胞提供了可能。
如上文所说,激光显微切割是一项自动化的样本预处理技术,这项技术能够在显微镜下从混合的细胞群体中分离出特定的细胞,而这种从复杂组织中分离纯化单个细胞的技术能够提高基因组分析的精确度。在显微镜下,通过细胞识别软件的处理,组织薄片中的细胞可被单个区分开,因此可以运用超强脉冲激光对组织直接进行切割。
近年来,激光显微切割技术的发展使激光切割的宽度能够少于1微米,因此目标细胞不会被激光束所影响,甚至活细胞也不会被激光束的切割所损伤,在适当操作的情况下,激光切割后的细胞仍然可以被用来克隆或重新培养[39]。激光显微切割技术常被用来从组织、血液甚至精子样本中分离少量细胞或单个细胞。这些细胞能够通过形态学、免疫组化染色、原位杂交的方法被选择且确定位置。
目前,已有大量研究使用了激光切割技术。2012年,华盛顿大学的Allen Jones和爱丁堡大学的Seth Grant共同发表了他们关于人类大脑转录组学的细胞图谱研究。他们将数字化的大脑分子图谱集成的方法引用到了模式生物上,利用激光切割的方法筛选出目标样本,发明了一种能够在样本中构建全面的转录组图谱的技术[40]。2014年,Allen大脑研究所的John Hohmann和Ed Lein利用激光显微切割技术,对妊娠中期的人类大脑样本构建了全面的转录组图谱,为人们了解大脑的发育过程提供了丰富的信息资源[41]。2014年,瑞典卡洛琳斯卡研究院的Carlos Ibanez和Sten Linnarsson将激光显微切割技术与大规模RNA测序相结合,对小鼠的内侧神经节突起进行了空间相关转录组分析,他们在中间神经元成熟的过程中发现了有明显差异的祖细胞群,这揭示了哺乳动物中枢神经系统的基因表达的空间异质性[42]。2016年,中科院上海细胞与生化研究所的景乃禾团队利用激光显微切割技术从小鼠的单个胚胎中分离出目标位置的细胞群,进行了单细胞RNA测序,揭示了小鼠胚胎原肠胚期的空间转录组信息和细胞身份[43]。
激光显微切割原理
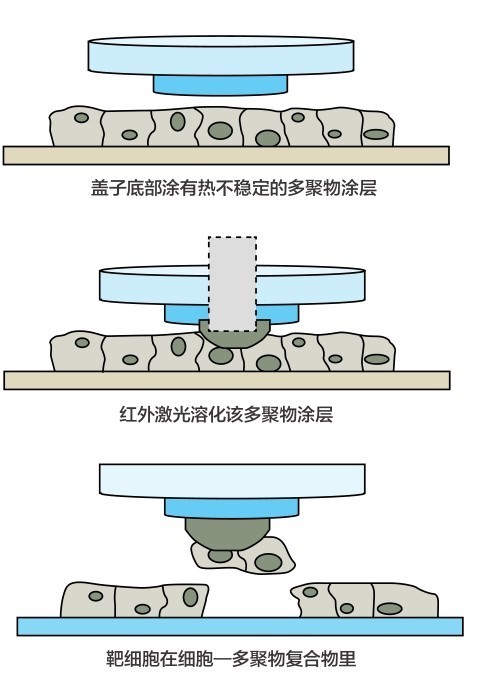
“古老”的原位荧光杂交技术也在单细胞化,并且与测序相结合。2014年,加州理工的Long Cai提出了荧光原位测序技术,它能够对还在组织或培养基中的细胞利用第二代测序直接进行测序。整个技术的基础是一种新型的核酸文库构建技术,这种技术能够在生物组织内进行稳定的交联c DNA扩增[44]。通过高强度的显微观测、生化处理、图像处理以及生物信息学分析,人们最终可以得到目标细胞的测序序列。2015年,哈佛大学的庄小威团队在《科学》杂志上发表了他们关于在单细胞中进行高度复用的空间解析RNA图谱的研究。他们发表了一种名叫MERFISH(多重抗误差原位荧光杂交)的技术,能够在单细胞中实现数千条RNA的拷贝数和空间定位的成像[45]。
荧光原位测序技术将空间相关的RNA-FISH技术和全转录组RNA图谱技术相结合,通过单个分子原位RNA定位来保持组织的形态。使用荧光原位测序,让分析不同空间分布或拷贝数变化的单个细胞转录组成为可能,这些分析能够协助描述组织中复杂的调解网络以及细胞类型的原位鉴定。
大多数的人体组织较厚,而且不透明。那么有没有一种方法让人们能够在保持组织原样的同时,对特定细胞进行定位和观察呢?2013年4月,斯坦福大学的Karl Deisseroth在《自然》杂志上发表的Clarity技术,可以使小鼠组织透明化[46]。通过Clarity,人们能够在器官中定位目标细胞的三维位置。Clarity的诞生使人们走进了器官图像分析的新纪元,改变了人们对器官内部的认知。

结合组织学和化学工程的工具,研究者们开发了一套不通过切割小鼠的大脑便能解析其三维复杂性和分子表达的方法。整个技术的核心是将小鼠大脑中的脂质置换成水凝胶聚合物。将小鼠的大脑放置于水凝胶单体的悬浮液后,大脑本身就会通过一种类似于石化的过程产生水凝胶聚合物,而且这种聚合物不会和脂质相结合。将脂质通过电泳过程移除之后,就会留下一个三维透明的大脑,且保留了所有的重要结构,如神经元、轴突、树突、突触等。
Clarity使人们能够对大脑进行完整的细节化的结构研究,对于了解健康和疾病状况下的大脑功能具有重要的意义。目前,Clarity已经被用于小鼠的大脑、胰腺、肾脏、肺、肠道和肝脏的研究中,但是对于具有很多非细胞基质组成的器官(如皮肤、齿龈等)的研究,Clarity仍具有局限性。
除了上述技术,神经科学研究者也将神经科学常用的电生理技术与测序相结合。膜片钳测序(Patch-Seq)在2015年由贝勒医学院和卡罗琳斯卡学院的Andreads Tolias和Rickard Sandberg共同发表在《自然·生物技术》杂志上[47]。在过去的几十年中,科学家们一直在利用一种叫作全细胞膜片钳的技术来测量神经细胞的电活动,比如神经元产生神经冲动时的独特的电位变化。然而,每个神经元之间的基因表达水平是不同的,之前并没有能够将单细胞的基因差异和膜片钳记录相结合的研究方法。在很长的一段时间内,想要研究神经元的细胞生理活动和基因表达之间的关系是一件很困难的事情。
Pathc-seq是一项将膜片钳记录与单细胞RNA测序相结合的技术,能够被用于同时研究单个神经元的形态学、生理学和基因表达图谱等。通过追踪电生理特征,研究者可以侦探到具有特点的目标细胞,随后用膜片钳吸管将细胞内容物吸出,进行RNA测序。对于神经细胞研究而言,Patch-Seq技术能够对目标细胞进行精确的内容物提取以研究神经元的多样性,对神经系统中复杂的细胞类型的分类研究有极大的帮助。
细胞图谱的意义重大,可以说是继人类基因组测序之后的又一个“兵家必争之地”。现在的局面是顶尖研究所三足鼎立:桑格研究所、布罗德研究所和新秀Biohub。
桑格研究所是一家位于英国的研究机构,其利用基因组测序技术来推动人类对生物和疾病的认知,以改善人类的健康。1993年10月4日,当时还被称为“桑格中心”的桑格研究所在英国创立,创立之初,整个机构仅有不到50名员工,如今所有园区的员工数量已经超过3000人。以建设大规模的世界级研究中心为目标,桑格研究所用20多年的时间,从最初的测序中心发展到基因组研究领域的行业领先机构。作为一家在遗传学领域世界领先的研究机构,桑格研究所旨在对人类病原生物学研究提供能够改变目前生物医学现状的思想[48]。
桑格研究所的细胞遗传学研究项目侧重于探索人类细胞中的基因组差异,以及在健康和疾病状况下的基因功能变化。他们实施了一个大规模的系统化基因筛选,目的是探索在自然状况和人工编辑状况下人类诱导多功能干细胞的基因变化,以及它们的分化衍生过程和其他细胞类型[49]。目前,这个项目正在研究参与感染、先天性免疫、代谢过程的细胞类别(如巨噬细胞、肝细胞、胰岛B细胞等),并且正在计划开展一项全面反映人体内细胞类别和功能的研究项目,以帮助人们更深入地了解、诊断、治疗、监测人类疾病。
细胞遗传学项目将使用来自已知健康状况的100多名受试者的多功能诱导性干细胞,使其分化为巨噬细胞、肝细胞、胰腺细胞等。通过分化后的细胞探索在宿主与病原之间的相互作用、先天性免疫反应、代谢反应过程中的细胞水平的变化。研究者们将来自细胞遗传学的结果和来自功能基因组学的数据相结合,利用创新型的算法来研究基因调控的机理,以帮助解释疾病之间的差异。利用单细胞研究的技术,科学家们计划建立一个全面反映人体内每个细胞的表观遗传学和转录组学的参考遗传图谱。同时,他们还计划开发一项基于Crispr-Cas系统来全面检测基因组层面的编码蛋白基因和长链非编码蛋白RNA的技术,以探索基因组成分对细胞表型的影响。这项计划将开发和改善一系列创新性的工具,以更加全面地分析单细胞研究的数据。
位于美国波士顿剑桥的布罗德研究所是细胞图谱计划的发起者之一。布罗德研究所起源于来自哈佛大学和麻省理工学院的科学家们数十年的非官方合作。1995年年初,来自麻省理工学院怀特海德研究所的科学家们意识到将遗传学应用于人类疾病研究的必要性,这促成了一些遗传性医学研究的初始项目,也促使哈佛大学和麻省理工学院在癌症和人类遗传学方面开拓新方法的科学家们形成了一套高效的合作网络[50]。随后,1998年哈佛医学院成立了化学与细胞生物研究所(ICCB),以帮助实现将化学基因组作为未来了解人类生物学和疾病的工具的目标。
这些项目说明了具有不同背景的研究者齐心协力解决分子医学问题中的重大挑战的重要性。由此可见,一个新型的正式的合作机构是十分必要的,它需要具有开放、合作、多学科交叉、能够组织任何规模的科研项目的特点。更重要的是,哈佛大学和麻省理工学院的遗传学家和生物化学家们能够互补合作,将基础的分子研究理论转化为对人类疾病的新型研究。2002—2003年,创始人依莱、埃德斯·布罗德和哈佛大学及附属医院、麻省理工学院、怀特海德研究所的研究者们勾勒出了这个新型研究机构的雏形。2003年,在依莱和埃德斯·布罗德的捐赠下,布罗德研究所正式宣告建设,并于2004年5月建成。截止到2014年,布罗德研究所总计获得超过了10亿美元的捐款,成为生物医学研究的领头者。
布罗德研究所的人类细胞图谱计划把来自世界各地的生物学家、临床医师、物理学家、计算机科学家、软件工程师和数学家们汇集起来。这些科学家们将各自不同的专业知识相互结合,为了一个共同的目标——建立全面的人类细胞遗传图谱而相互合作。只有建立了这个能够解析不同细胞类型的图谱,精确定位人体中的所有细胞,分析它们的基因表达水平,我们才能准确地描述所有的细胞活动,了解细胞网络结构。一个全面的细胞图谱使鉴定所有的细胞类类型(甚至亚型)、定位细胞的空间位置、区分不同的分化阶段和细胞状态成为可能;还使研究者能够追踪细胞谱系,比如追溯红细胞在骨髓中的干细胞来源。细胞图谱计划将会帮助鉴定不同疾病的生物标记物和各类特征,为各类疗法提供新的靶点目标,为人类生物学研究提供一个全新的视角[51]。
2016年,Facebook首席执行官扎克伯格和他的妻子陈丽霞捐赠6亿美元创立了Biohub,以推动加利福尼亚州湾区生物医学的合作和发展。这是扎克伯格及妻子投资的第一项科学慈善机构[52]。新秀Biohub将融合来自加州大学伯克利分校、斯坦福大学和加州大学三藩分校的科学家们来推动人类疾病的研究。Biohub的创立目标是帮助治愈、预防、操控人的一生中所有的疾病,创立未来生命科学研究的新蓝图。所有Biohub的科学家、研究院和工程师们将尝试破解人类疾病的复杂性,并为治愈疾病提供新的方法。除了合作和科学研究之外,Biohub的另一个使命是培养青年科学家成为行业的领头人。
扎克伯格和他的妻子陈丽霞将细胞图谱研究作为其30亿美元医疗研究捐赠的首个目标。人类细胞的未解之谜是很多疾病发生的根源, Biohub的细胞图谱项目将通过研究健康人类中细胞工作的方式,尝试揭开这些谜团。其研究的重点更加侧重于在疾病发生时这些细胞发生的反应,以描述细胞在疾病刺激下的内部机理变化[53]。
让我们期待细胞图谱为医学科学带来新的突破!
专家点评
徐迅
华大基因研究院院长,国家基因库执行主任。
细胞是组成生命的最基本单位。人体细胞究竟有多少种类,不同种类的细胞如何实现不同的功能,当疾病发生时这些细胞发生了什么样的改变?现有的知识将细胞分为400多种,但究竟有多少种谁也说不清楚。随着单细胞技术,尤其是大规模单细胞测序研究的开展,越来越多的新的细胞亚型被鉴定出来。人体细胞图谱计划试图在基因表达水平精确地定义人体的每一个细胞,如同人类基因组计划那样全面透彻地解读人体细胞“天书”。
第一个单细胞转录组研究是采用微阵列芯片技术完成的,而第二代测序技术的出现使转录组研究进入了一个被称为“RNA测序”的阶段。从2009年至2017年将近8年的时间里,单细胞转录组技术飞速发展,特别是基于纳米微升的droplet技术将单细胞RNA测序的成本降低到一个市场可以接受的水平。就是在这样的背景下,桑格研究所和布罗德研究所牵头发起了“人体单细胞图谱”计划,并且获得了Chan Zuckerberg Initiative基金会的大力支持。据悉,第一批资助计划将很快进入实际申请阶段。
尽管人体单细胞图谱计划还处在孕育的早期阶段,但我们已能触摸到即将带来的巨大变革,而这个变革将不亚于人类基因组计划。首先,疾病的诊断模式将迎来全新的飞跃。人体单细胞图谱提供了健康人的完整细胞目录,而疾病细胞通过单细胞RNA测序找到的与“已知目录”的差异信息将成为疾病诊断的重要线索,最终迎来一个“人体疾病细胞图谱”,让疾病能够在更早期就被诊断出来。其次,药物研发的速度将加快而成本将下降。对于已知靶点的药物,借助人体单细胞图谱的信息,将更容易通过大数据方式预测该药物的副作用,甚至针对特定病人精确地预测其是否有可能出现严重的诸如肝肾功能衰竭等副作用。
人体单细胞图谱是一个极其“大胆的”计划,想要切实落实并最终获得高质量的图谱,仍面临许多挑战。众多的世界顶尖的实验室将加入其中,如何建立标准化的操作流程,使来自不同实验室的数据可以有意义地被整合,是首当其冲需要解决的问题。
专家点评
曹虎
科特勒咨询集团(KMG)中国区总裁。
“如果你想用一个世纪的时间来寻找治疗疾病的方法,最好先把重点放在基础研究上,因为你根本无法预测这些治疗方法从何而来!”科学慈善联盟主席(Science Philanthropy Alliance) Marc Kastner在给CZ Biohub的建议中如是说。而且Chan 和Zukerberg接受了这个建议,并且制定了一个宏伟的计划:人类细胞图谱(Te Cell Atlas),旨在对人体内的每一种细胞进行单细胞测序,描绘其分子特征。作为一个开源的项目,人类细胞图谱产出的数据将向所有研究者开放。它显然将对新药的研发、新治疗手段的产生发挥重要的促进作用,同时在这个项目的开展过程中产生的新技术很可能开启很多基础研究和临床研究的新篇章。
从技术上看,今天的单细胞测序技术仍然面临着至少4个方面的挑战:有效的单细胞分离;基因组扩增;测序价格;有效的数据解读。虽然单细胞测序已经在辅助生殖领域进入了初步的临床应用阶段,但是目前的数据准确性、检测周期和价格都还是临床推广的瓶颈。CZ Biohub的联席主席斯蒂芬·夸克(Stephen Quake)显然是这个领域的翘楚。夸克将利用细胞微流体进行单细胞分离;利用超快、高效的测序仪,极大地降低测序成本、极快地提升测序速度;使用基因组编辑(如CRISPR)进行单细胞的标记和追踪技术,基于基因活动来定位人体器官中的各种细胞。相信这些技术将在项目的推进过程中逐渐迭代,将来很有可能发展成适合在临床开展的临床检测项目,满足目前无法满足的临床需求。比如目前在肿瘤的液体活检领域,CTC(循环肿瘤细胞)已经作为成熟的检测手段用于肿瘤的复发监控。但是目前还没有办法分析CTC的基因组,了解其异质性并针对其突变情况精准用药。而用于人类细胞图谱的技术将满足这个临床的需求。再如,目前很多的肿瘤患者无法判断其原发灶的组织来源,通过对肿瘤患者进行单细胞测序,检索人类细胞图谱,临床医生将很容易判断肿瘤细胞的来源并制定更精准的治疗方案。在遗传疾病领域,我们已经知道很多遗传疾病存在基因嵌合的现象。夸克位于斯坦福大学的实验室最近就发现了由于SCN5A基因嵌合导致的长QT 综合征。
正如17年前的人类基因组计划催生了今天正在蓬勃发展的精准医疗产业,人类细胞图谱必将把基础研究、新药研发、临床研究和分子诊断带入一个全新的境界。
专家点评
田埂
元码基因联合创始人,曾任清华大学基因组与合成生物学中心主管,华大基因华北区第一负责人,天津华大创始人、总经理,深圳华大基因研究院研发副主管。
意义堪比人类基因组计划的人类细胞图谱项目,将破译出人体中每个细胞的类型和特性,构建健康人体的参考图。人类细胞图谱是生命科学领域很长一段时期以来最为振奋人心的提案。对于健康和疾病而言,细胞是生命最基础的组成部分,只有对细胞有清晰的了解,才能够了解病理机制,提供有效的治疗方案。这一项目的完成对整个人类来说无疑具有非常重大的意义,它使人类向真正的“个性化医疗”时代又迈进了一步。
细胞图谱项目是医学上一场革命的开始,但这场革命的成功将需要更长的时间。有可能“理想很丰满,但现实却很骨感”。单从技术层面上看,不断发展的科学技术完全可以支持细胞图谱绘制的实现,如细胞微流体、高效的测序仪和先进的标记及染色技术等。从经济角度上看,现在的测序成本已经低至1个细胞6美分,人类对自身研究的需求远远可以让这个价格显得微不足道。
细胞图谱是一项基础工程,对于这项技术的复杂度和工作量,不同的科学家在认识上还存在着一些分歧。有的科学家认为人体内细胞的种类和数目繁多,有的还存在众多亚型,使得人类细胞图谱项目比人类基因组计划有更多的未知难度和更大的工作量。对此我们则持乐观的态度,“莫道浮云遮蔽日,严冬过尽绽春蕾”,实验科学永远要在做了之后才能真正地挖掘出意义。细胞图谱对于科学体系与细胞机制的推动会在未来百年之内不断发酵和沉淀。我们相信,通过完成这项有着革命性意义的工作,会让人类细胞图谱项目成为21世纪最耀眼的注脚之一。
